0 引言
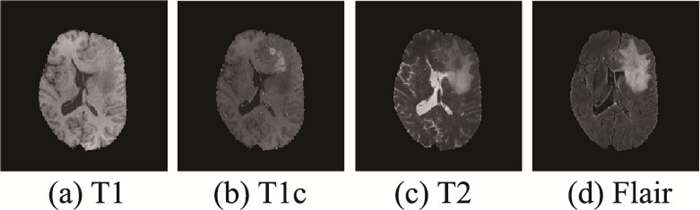
脑肿瘤是在颅内异常生长的细胞群,时刻威胁着患者的生命[1]。脑肿瘤的形状不规则,且生长位置不定等特性给脑肿瘤分割任务带来巨大的挑战[2]。医学成像技术向临床医生提供了更多准确可靠的信息[3]。其中,核磁共振成像(magnetic resonance imaging,MRI)因其具备非入侵性、多序列成像等特点被广泛应用于肿瘤患者病情的诊断[4]。MRI主要的成像方式有4种,如图 1所示,依次为T1(T1-weighted)、T1c(contrast-enhanced T1-weighted)、T2(T2-weighted)和Flair(fluid attenuated inversion recovery)[5],可为医生提供同一部位不同方向的脑肿瘤成像信息。医生手动分割脑肿瘤既费时又受主观意识的影响[6]。因此,研究一种高效准确的全自动脑肿瘤分割方法具有重要临床价值。
|
| 图 1 4种脑肿瘤MRI成像模态图 Fig.1 Four brain tumor MRI imaging modalities |
近年来,卷积神经网络(convolutional neural network,CNN)被广泛应用于脑肿瘤图像分割任务,并取得不错的分割效果[7]。目前脑肿瘤分割网络主要分为2维CNN网络和3维CNN网络。其中,2维CNN网络从3维图像中提取2维的切片,在提高计算效率的同时缓解了数据不平衡的影响,如FCN[8]、U-Net[9]等。FCN使用1×1卷积代替全连接层,对肿瘤图像的每个像素进行了预测,实现了像素级别的分类。U-Net在FCN的基础上提出高效的编—解码结构,并利用跳跃连接将编—解码路径提取的特征图进行合并,获得不错的脑肿瘤分割结果。2维CNN网络虽然获得不错的分割精度,但网络的输入是2维切片,对3维MRI图像提取的信息较为片面,且分割后的切片合成3维分割结果时,会出现混叠、断层等问题。
3维CNN网络避免了2维CNN网络的缺陷,可以直接处理3维的图像数据,如Çiçek等[10]针对2维网络分割结果出现的锯齿、断层等问题,利用3维卷积核将2维U-Net网络拓展到3维空间中,直接处理整张3维图像,获取了丰富的特征信息,并加快了特征学习的效率。Jiang等[11]采用两个模型级联的策略来缓解脑肿瘤数据类别不平衡的问题。第一个U-Net用来筛除大量的背景信息,获取初步的脑肿瘤分割图送入第二个网络,然后第二个网络对初步的分割图进行细化分割,获得了较精准的分割性能。目前现有的3维脑肿瘤分割方法虽然取得了较高的分割精度,但随着网络深度的不断加深,网络参数和计算量的增加限制了其在显存较小的硬件平台上的运行。
轻量级网络通过设计更加高效的结构,在保证分割精度的同时大幅度降低网络参数量。如Chen等[12]以组卷积结合多尺寸的空洞卷积捕捉图像不同尺度空间上的相关性,有效缓解了脑肿瘤各子区域占比不平衡对分割精度的影响,但过多的使用组卷积带来了通道间信息交流不畅的问题。Luo等[13]提出一种轻巧的分层解耦卷积网络,从多视图上提取图像的特征信息,提高了网络对空间上下文信息的感知能力,但过于轻量化的网络结构带来了分割性能的下降。
目前现有的轻量级网络虽然降低了网络参数量和计算量,但在分割精度上仍有较大提升空间。为此,本文提出一种基于ShuffleNet的高效脑肿瘤分割网络,该网络在U-Net的基础上使用Shuffle结合深度可分离卷积以缩减网络的参数量和计算量;同时在有限的GPU内存空间上设置较深的卷积层数以最大化网络的非线性提取能力,并利用多层并行的卷积层增加网络的多尺度表示能力,实现了分割性能和分割速度的平衡。最后在测试集上进行了实验验证。
1 网络结构2018年,Ma等[14]提出更为高效的ShuffleNet轻量级结构,本文将ShuffleNet的核心模块Shuffle扩充至3D版作为U型网络的基本结构,在有限的GPU内存空间上搭建最大化的U型网络。为提升网络多尺度信息的提取能力,引入空洞空间金字塔(dilated spatial pyramid,DSP)池化模块以及加入混合感受野的Shuffle(Shuffle with mixed receptive fields,Mix-Shuffle)单元。为了让网络得到更好的训练,使用Dice和Focal损失函数加权相加的混合损失函数训练网络,最终得到基于ShuffleNet的多尺度高效脑肿瘤分割网络(multi-scale efficient brain tumor segmentation network based on ShuffleNet,MSEV-Net)。
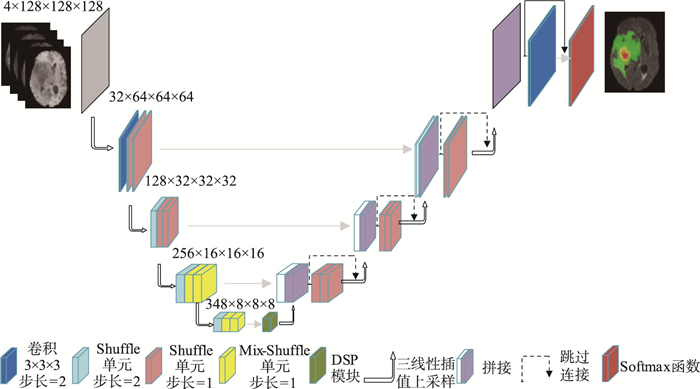
1.1 网络整体框架MSEV-Net完整网络框架如图 2所示,整体分为编码阶段和解码阶段两部分,编码阶段分为4层,每层由一个步长为2、核大小为3×3×3的Mix-Shuffle单元和两个相同核尺寸、步长为1的Mix-Shuffle单元组成,前者用来替代池化层降低图像分辨率,后者用来学习图像的特征信息,编码器底层的DSP模块用来将提取的多尺度信息进行融合。解码阶段同为4层,每层由反卷积、Shuffle单元以及残差结构组成,用于恢复图像的分辨率,还原图像的细节信息。另外,在每次上采样后,会通过跳跃连接将编码阶段提取的语义信息与采样复原后的细节信息进行拼接,实现对目标分割区域的精准定位与识别。最后,使用Softmax函数将输出特征图转化为与输入图像同尺寸的概率矩阵。
|
| 图 2 MSEV-Net结构 Fig.2 The structure of MSEV-Net |
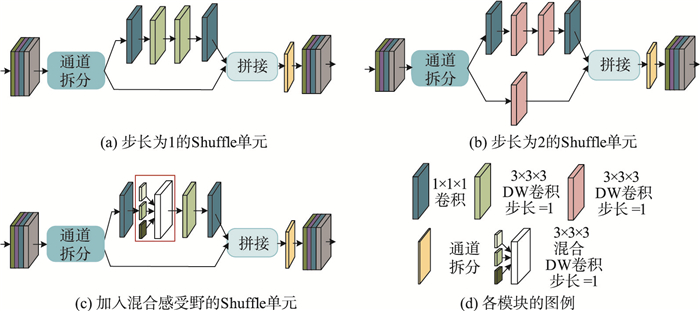
Shuffle单元详细网络结构如图 3所示,其中,图 3(a)是基础的Shuffle单元,步长为1,由通道拆分(channel split)[14]、深度可分离卷积(depthwise separable convolution)[15]和通道混洗单元(channel Shuffle unit)[16]三个部分组成;图 3(b)是步长为2的Shuffle单元,用来替代池化层,压缩图像的分辨率;图 3(c)是加入混合感受野的Shuffle单元,在深度可分离的操作过程中采用多分支结构,从上至下采用空洞率递增的空洞卷积。
|
| 图 3 Shuffle单元结构图 Fig.3 The structure diagram of shuffle unit |
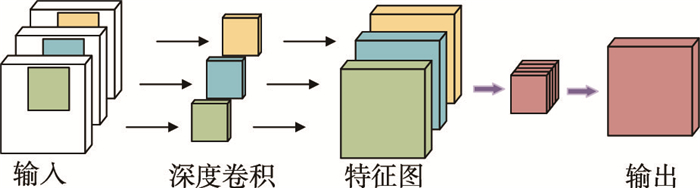
深度可分离卷积如图 4所示,由深度卷积(depth-wise convolution,DC)和逐点卷积(point-wise convolution,PC)两部分组成,相比于标准卷积,其参数量和运算成本比较低,能够大大简化网络的复杂度。深度卷积用于提取图像的空间特征信息,其参数量和计算量为
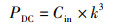
|
(1) |

|
(2) |
|
| 图 4 深度可分离卷积 Fig.4 Depth-wise separable convolution |
式中,Cin表示输入特征图的通道数,PDC、FDC分别表示深度卷积的参数量、计算量,k表示卷积核的大小,D×H×W表示特征块尺寸。深度卷积输出与输入的通道数相同,无法拓展输出图像的深度,且割裂了不同通道内相同位置上的信息交流。因此需要使用逐点卷积来促进通道间的信息交流,逐点卷积的参数量和计算量为
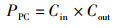
|
(3) |
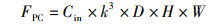
|
(4) |
式中,Cout为输出特征的通道数,PPC、FPC分别表示逐点卷积的参数量、计算量。深度可分离卷积与标准卷积的参数量和计算量之比为
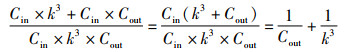
|
(5) |
由式(5)可知,深度可分离卷积将空间维度与通道维度分开处理,使得计算由k3与Cout的相乘转化为相加,从而大大降低了参数量和计算量,因此本文的编码结构提高了计算效率并节省了GPU的内存资源。
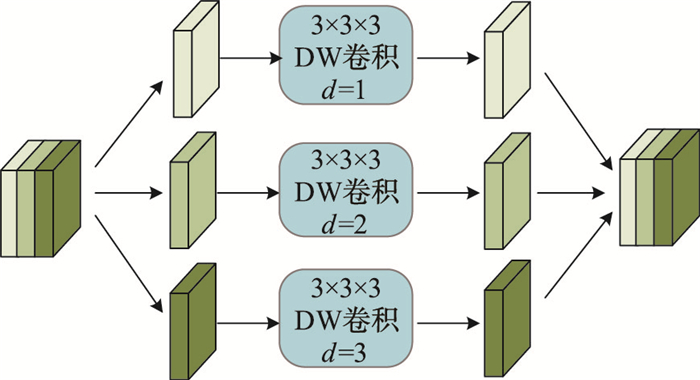
另外,为进一步提升网络的特征提取能力,本文提出采用混合感受野的深度可分离卷积,如图 5所示,将输入通道划分为3组,通过设置不同的空洞率获取多尺寸的感受野,其中小尺寸感受野提取图像的细节信息,大尺寸感受野提取图像的轮廓信息。然后将不同感受野获取的特征图相加融合,充分获取图像的上下文信息。此外,由式(5)可知,当Cout较大时,深度可分离卷积的参数量主要集中在PC部分上,即使DC部分采用混合感受野结构,也不会导致参数量大幅度增加。因此,本文在编码阶段底层使用加入混合感受野的Mix-Shuffle单元,避免网络参数量骤增。
|
| 图 5 混合感受野的结构 Fig.5 The structure of mixed receptive fields |
本文在Shuffle单元中引入通道拆分操作,将输入通道均分成上下两路,其中上路分支进行连续卷积提取抽象信息,下路分支进行同等映射,最终采用通道拼接的方式合并两路信息。双路径结构能提取不同的特征信息,并以通道拼接的方式增加输出特征的多样性,但该结构割裂了上下两路径通道间的信息交流。因此,本文在通道拼接后引入通道混洗单元,以促进分支通道之间的信息交流。假设卷积层输出特征图有n个通道,n=c×d(c表示分组数)。通道混洗单元将特征图的通道维度重塑为(c,d),然后将其进行转置,最后再展平为c×d个通道。保证进入网络下个卷积层的特征来自不同的分组,以此加强通道间的信息交流,提升网络的分割性能。
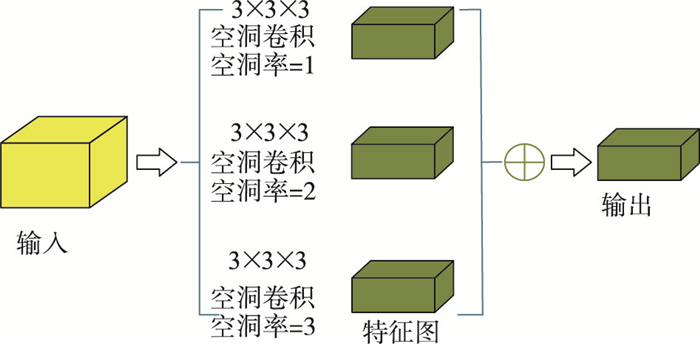
1.3 空洞空间金字塔池化经过4层的下采样已经将图像的分辨率压缩到8×8×8,此时已提取到高度抽象的特征信息,继续增加网络的深度进行下采样,在上采样复原图像时会在一定程度上导致图像信息的丢失,影响分割精度。为了堆叠出更深层次的网络并且避免采样导致的图像信息丢失,在底层加入空洞空间金字塔(dilated spatial pyramid,DSP)池化模块。DSP模块采用空洞卷积[17]代替普通的池化操作,使用空洞卷积可以得到输入与输出同尺寸的特征图,从而避免图像分辨率改变导致的信息丢失问题。DSP的结构如图 6所示,其采用多个卷积层并联的结构提取图像特征信息,每层卷积层有256个卷积核,且不同支路使用的空洞率不同。因此该模块拥有多个尺寸的卷积核来获取更密集的图像特征信息,最后使用逐元素相加融合特征信息,完成肿瘤分割区域的精准定位。
|
| 图 6 空洞空间金字塔池化 Fig.6 Dilated spatial pyramid pooling |
在脑肿瘤MRI图像中,不仅存在极端的背景和肿瘤前景的数据类别不平衡问题,而且在肿瘤的不同子区域之间也存在严重的数据类别不平衡的问题,本文旨在设计一款更优的损失函数,以缓解数据集中存在的类别不平衡问题,提升小目标区域的分割性能。
Focal损失函数是专为解决类别不平衡和训练中分类困难的问题而设计的。在训练阶段,该函数通过调节参数抑制易分类样本的权重,同时使网络聚焦于难分类样本。其表达式为
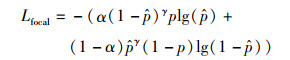
|
(6) |
式中,p为真实类别,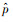
Dice损失函数是脑肿瘤图像分割领域最为常见的损失函数,具有较快的收敛速度和较好的性能。其表达式为
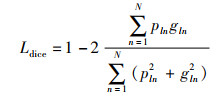
|
(7) |
式中,gln为类别l第n个体素的真值,pln为对应的预测值,N是样本中体素的总数。
Dice损失函数在脑肿瘤分割中比较常见,但受类别不平衡的影响导致稳定性差,而Focal损失函数可以改善类别不平衡问题。因此,本文将两种损失函数进行加权获得一种新的混合损失函数,该损失函数可以缓解类别不平衡对Dice损失函数分割效果的影响,从而获得更好的分割性能。其函数表达式如下:
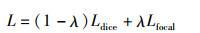
|
(8) |
式中,λ为加权系数,是当前迭代次数与总迭代次数的比值。
1.5 评价标准脑肿瘤分割算法的有效性主要通过比较网络的复杂度和分割精度来评估,本文通过Dice相似系数(dice similarity coefficient,Dice)和豪斯多夫距离(Hausdorff distance,Hausdorff)两个指标来评估脑肿瘤分割结果;本文通过参数量和计算量来衡量网络的复杂度[18]。
2 实验与结果分析 2.1 实验数据集与参数设置本实验采用BraTS2019[19]比赛官方公布的脑肿瘤数据库,其中训练集由HGG病例样本(225例)和LGG病例样本(75例)组成,且每个样本包含4张MRI序列图(T1,T1ce,T2,Flair)和1张真值标签图,所有图像的尺寸均为240×240×155。真值标签图由专业医师手动标注,标注包括增强肿瘤(ET-标签4),水肿(ED-标签2)以及坏死和非增强肿瘤(NCR/NET-标签1)。实验的验证集采用125个无标签患者病例,将分割结果上传到比赛在线评估服务器上进行评估,然后根据评估结果调试网络。最后,本文从最新公开数据集BraTS2021中新增的临床病人中随机抽选98个样本作为测试集,在测试集上测试所提网络在临床上的分割性能。
本文实验的代码使用Python 3. 8完成,服务器运行的系统环境为Ubuntu 16. 04,CPU为Intel® Core i9-9900X(3. 5 HGz),显卡为11GB的Nvidia GTX2080Ti,所有的实验网络使用1.9.0版本的PyTorch框架搭建。实验参数设置如下:网络输入图像块大小为128×128×128,批处理量设定为8,训练总轮数设定为500。使用Adam优化器对网络进行优化,初始学习率设定为1×10-3,初始衰减率设定为1×10-5。在网络训练阶段使用以下数据增强技术扩充训练集:1) 以0. 5的概率随机在轴向、冠状、矢状等方向镜像翻转;2) 在[-10°,10°]范围内随机旋转。
2.2 实验结果分析 2.2.1 不同损失函数下的网络分割结果为了验证改进的加权混合损失函数对分割结果的影响,实验分别使用不同的损失函数对MSEV-Net进行训练,并在BraTS2019验证集上进行验证评估,分割结果如表 1所示。ET表示增强肿瘤,WT表示全肿瘤,TC表示肿瘤核心。
| 损失函数 | Dice得分/% | ||
| ET | WT | TC | |
| Dice | 77.30 | 88.76 | 77.93 |
| Focal | 74.84 | 89.21 | 76.93 |
| Focal+Dice | 76.92 | 89.44 | 80.59 |
| 本文 | 78.25 | 89.51 | 81.60 |
由表 1中数据分析可得,本文提出的加权混合损失函数训练的网络分割效果最佳。与Dice和Focal直接相加的混合损失函数训练的网络相比,在所有分割性能指标方面均有提升,说明能更好地结合Dice和Focal的优点,解决数据类别不平衡问题。
2.2.2 DSP模块的消融实验为了比较DSP模块对分割性能的影响,设计了一系列不同构成的DSP模块的对比实验,具体的构造如表 2所示。在表 2中列出5种不同空洞率的实验方法。例如(1,2,2)表示方法2中有3个空洞卷积层,空洞率分别为1,2,2。方法1有1个空洞率为1的空洞卷积,即常规卷积。
| 方法 | 空洞率 |
| 1 | 常规卷积(d=1) |
| 2 | (1,2,2) |
| 3 | (1,2,4) |
| 4 | (1,2,3) |
| 5 | (2,4,6) |
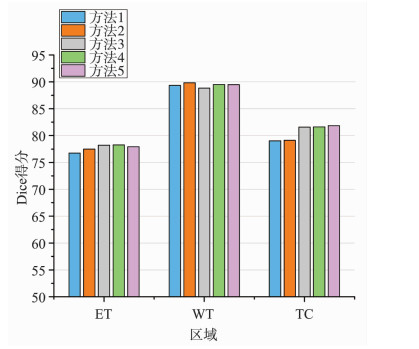
图 7和表 3显示了不同DSP模块的分割结果。从图 7和表 3的结果中可以看出,加入DSP模块可以有效提升模型的分割性能。方法2、3、4是探究组合增加对方式模型的分割效果的提升,根据结果表明,发现空洞率倍增的DSP模块分割效果不如递增的稳定。方法3、5是研究DSP模块空洞率的大小对模型分割性能的影响,根据结果表明,具有大空洞率的DSP模块整体的分割效果更佳。另外,根据实验结果表明,方法4在ET、WT、TC区域上取得了78. 25%、89. 51%、81. 60%的最佳结果。
|
| 图 7 不同DSP模块的分割结果 Fig.7 Segmentation results of different DSP modules |
| 方法 | Dice得分/% | ||
| ET | WT | TC | |
| 方法1 | 76.75 | 89.36 | 79.02 |
| 方法2 | 77.50 | 89.83 | 79.12 |
| 方法3 | 78.21 | 88.85 | 81.57 |
| 方法4 | 78.25 | 89.51 | 81.60 |
| 方法5 | 77.91 | 89.47 | 81.84 |
本文以Shuffle单元构建的U-Net为基准网络,在相同的实验环境下,验证不同模块的有效性。结果如表 4所示,其中Shuffle代表基准网络,DSP代表空洞特征金字塔池化,Mix-Shuffle代表加入混合感受野的网络。可以看出,分别加入DSP和混合感受野后,网络性能在小目标肿瘤ET上的分割效果有较为明显的提高。最后将两个模块同时加入获得最好的分割结果,其中ET小目标肿瘤分割精度提升2.47%。说明本文提出的模块可以提升小目标肿瘤ET的分割精度。
| 网络模型 | Dice得分/% | ||
| ET | WT | TC | |
| Shuffle | 75.78 | 89.47 | 81.06 |
| Shuffle+DSP | 77.09 | 89.59 | 81.00 |
| Mix-Shuffle | 77.26 | 89.38 | 81.52 |
| Mix-Shuffle+DSP | 78.25 | 89.51 | 81.60 |
图 8为基准网络加入不同模块后的分割结果。图中红色标注表示坏死和非增强肿瘤、绿色标注表示水肿、黄色标注表示增强肿瘤。从图中可以看出,4个网络的在水肿区的分割结果与真值标签较为接近。但在小目标肿瘤的精准分割方面,均不如两种模块同时加入后的网络。同时加入DSP和混合感受野后,网络降低了坏死区和增强肿瘤区的错误分割概率,对小目标肿瘤轮廓信息的识别更加细腻。
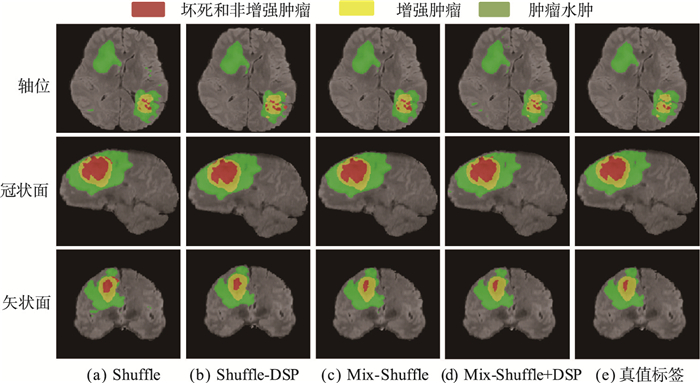
|
| 图 8 加入不同模块的分割结果 Fig.8 Segmentation results after adding different modules |
为了比较本文所提网络与其他较为先进的分割网络的差距,在BraTS2019的验证集上,将所提的MSEV-Net网络与其他5种先进网络进行了对比实验,所选对比网络为:经典3D-UNet[10]、轻量级3D-ESPNet[20]、轻量级DMF-Net[12]以及两个目前处理BraTS2019数据集最新的网络Mickinley[21]和Milan[22]。所有网络实验结果如表 5所示。
| 网络模型 | 参数量 | 计算量 | Dice得分/% | Hausdorff /mm | |||||||
| ET | WT | TC | AVG | ET | WT | TC | AVG | ||||
| 3D-UNet[10] | 1.621×107 | 1.669 53×1012 | 73.70 | 89.43 | 80.70 | 81.28 | 6.41 | 12.3 | 10.44 | 9.72 | |
| 3D-ESPNet[20] | 3.36×106 | 7.651×1010 | 66.32 | 87.17 | 78.62 | 77.37 | 6.84 | 7.42 | 9.74 | 8.00 | |
| DMF-Net[12] | 3.88×106 | 2.704×1010 | 77.60 | 90.00 | 81.50 | 83.03 | 2.99 | 4.64 | 6.22 | 4.62 | |
| Mckinley, et al[21] | 75.00 | 91.00 | 81.00 | 82.33 | 4.35 | 5.97 | 6.21 | 5.51 | |||
| Milan, et al[22] | 76.33 | 90.02 | 79.68 | 82.01 | 3.89 | 5.72 | 6.97 | 5.53 | |||
| MSEV-Net(ours) | 6.51×106 | 1.907×1010 | 78.25 | 89.51 | 81.60 | 83.12 | 2.94 | 5.27 | 6.57 | 4.93 | |
由表 5可知,本文提出的MSEV-Net网络平均Dice指标最高,平均Hausdorff距离指标排名第二。在分割精度方面,相较于次优网络DMF-Net,MSEV-Net在增强肿瘤区域Dice指标提升0. 65%。另外,MSEV-Net除WT的Dice指标较低外,其余Dice指标均优于其他模型,尤其是增强肿瘤区域,说明本文所提网络在脑肿瘤分割任务中具有不错的分割精度,并对增强肿瘤区域有更优异的分割表现。在模型复杂度方面,相较于3D-UNet,参数量和计算量大幅度降低,且与轻量级网络DMF-Net相比,网络的FLOPs降低29. 47%,说明本文所提网络具有较低的参数量和拥有较快的计算速度。
不同网络对脑肿瘤图像分割的可视化结果如图 9所示。从图中可以看出,采用U-Net网络分割的WT、ET区域(图 9(b))出现大块的假阴性区域,即有大块病变区域未能检测出。DMF网络(图 9(c))能够分割出大致的肿瘤轮廓,但在小目标肿瘤ET区域的分割结果与真值差距较大。MSEV-Net的分割结果割如图 7(d)所示,与其他网络相比,分割的肿瘤区域与真值标签相似度最高,尤其是对ET区域的分割,周围零星的病变区域也被进一步挖掘,表明本文训练的网络有效地改善了小目标肿瘤区域的分割结果。
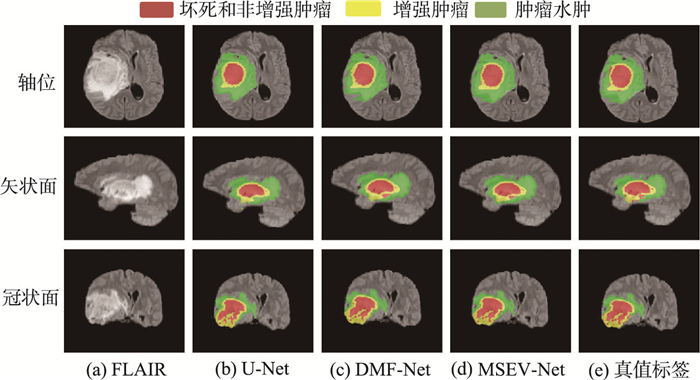
|
| 图 9 不同网络对脑肿瘤图像的分割结果 Fig.9 Segmentation results of brain tumor images by different models |
综上所述,本文提出的MSEV-Net网络对ET、TC脑肿瘤区域更加敏感。原因是混合损失函数训练的网络更优以及DSP模块提升了网络对图像多尺度特征的提取能力。同时,本文以较低的参数量实现了深层次的U型网络结构,并获得较高的运行速度。
2.2.5 测试集上的分割结果为验证所提网络在临床中的分割性能,本文在最新公开的数据集BraTS2021新增的临床病例中选取98个样本作为测试集,在该测试集上评估本文网络在临床中的分割性能。并与U-Net和Shuffle单元构建的基准网络进行对比,结果如表 6所示。
| 网络模型 | Dice得分/% | ||
| ET | WT | TC | |
| U-Net | 79.10 | 88.64 | 81.32 |
| Shuffle | 83.23 | 89.55 | 86.94 |
| 本文 | 85.10 | 90.82 | 86.89 |
从表 6可以看出,U-Net在测试集上的临床效果最低,使用Shuffle单元构建的U-Net在3个肿瘤区域的Dice表现均有提升,尤其在肿瘤核心区域和增强肿瘤区域,Dice值提升超过4%。本文所提方法与Shuffle相比,在肿瘤核心区域上的分割较为接近,在增强肿瘤区域和整体肿瘤区域的分割结果分别提升1. 87%和1. 27%。结果表明本文所提网络在临床上的分割效果更好,尤其是在肿瘤核心区域上的分割效果。
图 10显示了不同模型在测试集上的分割结果。由图可以看出,U-Net的分割效果较差,在非肿瘤区域出现了大面积的假阳性区域,即正常细胞错误判决为癌变细胞。Shuffle和本文网络整体分割效果较为接近,但本文网络MSEV-Net对坏死区域和增强肿瘤区域的边界识别更加准确,对小目标肿瘤的分割结果更接近真值标签。
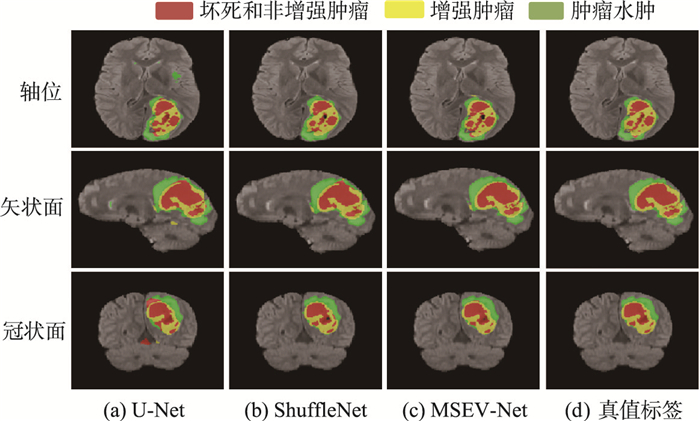
|
| 图 10 不同模型在测试集上的分割结果 Fig.10 Segmentation results of different models on the test set |
目前现有的轻量级脑肿瘤分割网络虽然简化了自身的结构,但脑肿瘤尤其是小目标肿瘤区域分割精度偏低。对此,本文提出一种基于ShuffleNet的多尺度高效脑肿瘤分割网络——MSEV-Net,并使用改进的加权混合损失函数训练网络,在实现小目标肿瘤区域高精度分割的同时保证网络的轻量化。本文网络在验证集上肿瘤增强区、全肿瘤区和肿瘤核心区的Dice值分别为78. 25%、89. 51%和81. 60%,在测试集上相应的Dice值为85. 10%,90. 82%,86. 89%,参数量与浮点数运算次数分别为6. 51 M和19. 07 G,与BraTS2019数据集上的其他网络相比,本文网络具有分割准确度高、网络复杂度低等优点。由此可知,3维网络可以采用轻量级卷积方式来最大化加深网络的深度,在保证网络较轻量的同时可获得更好的分割精度。目前,实验测试集和训练集中高级胶质瘤患者的病例样本占比较高,低级胶质瘤患者的病例比较稀少,未来将收集更多的脑肿瘤患者病例样本进行特征训练。另外,设计所提网络未考虑Shuffle V2所提的四条准则,下一步将根据这四条准则进一步优化本文所提网络。
| [1] |
Sumbal R, Kanwal R, Muhammad S H A. An insight into the risk factors of brain tumors and their therapeutic interventions[J]. Biomedicine & Pharmacotherapy, 2021. DOI:10.1016/j.biojha.2021.11269 |
| [2] |
Menze B H, Jakab A, Bauer S, et al. The multimodal brain tumor image segmentation benchmark (BRATS)[J]. IEEE Transactions on Medical Imaging, 2015, 34(10): 1993-2024. DOI:10.1109/TMI.2014.2377694 |
| [3] |
徐军, 刘慧, 尹义龙. 基于非局部自回归学习的医学图像超分辨重建方法[J]. 模式识别与人工智能, 2017, 170(8): 747-753. Xu J, Liu H, Yin Y L. Medical image super-resolution reconstruction method based on non-local autoregressive learning[J]. Pattern Recognition and Artificial Intelligence, 2017, 170(8): 747-753. |
| [4] |
李仲年, 张涛, 张道强. 基于自监督边缘融合网络的MRI影像重建[J]. 模式识别与人工智能, 2021, 214(4): 361-366. Li Z N, Zhang T, Zhang D Q. Self-supervised edge-fusion network for MRI reconstruction[J]. Pattern Recognition and Artificial Intelligence, 2021, 214(4): 361-366. |
| [5] |
Liu Y J, Du J, Yue G H, et al. Scale-adaptive super-feature based metricunet for brain tumor segmentation[J]. Biomedical Signal Processing and Control, 2022. DOI:10.1016/j.bspc.2021.103442 |
| [6] |
张恒良, 李锵, 关欣. 一种改进的3维双路径脑肿瘤图像分割网络[J]. 光学学报, 2021, 41(3): 60-67. Zhang H L, Li Q, Guan X. An improved three-dimensional dual-path brain tumor segmentation network[J]. Acta Optica Sinica, 2021, 41(3): 60-67. |
| [7] |
高程玲, 叶海良, 曹飞龙. 基于三重交互关注网络的医学图像分割算法[J]. 模式识别与人工智能, 2021, 215(5): 398-406. Gao C L, Ye H L, Cao F L. Medical image segmentation via triplet interactive attention network[J]. Pattern Recognition and Artificial Intelligence, 2021, 215(5): 398-406. |
| [8] |
Evan S, Jonathan L, Trevor D. Fully convolutional networks for semantic segmentation[J]. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2017, 39(4): 640-651. DOI:10.1109/TPAMI.2016.2572683 |
| [9] |
Ronneberger O, Fischer P, Brox T. U-Net: Convolutional networks for biomedical image segmentation[M]//Lecture Notes in Computer Science, vol. 9351. Berlin, Germany: Springer, 2015: 234-241.
|
| [10] |
Çiçek Ö, Abdulkadir A, Lienkamp S S, et al. 3D U-Net: Learning dense volumetric segmentation from sparse annotation[M]//Lecture Notes in Computer Science, vol. 9901. Berlin, Germany: Springer, 2016: 424-432.
|
| [11] |
Jiang Z Y, Ding C X, Liu M F, et al. Two-stage cascaded U-Net: 1st place solution to brats challenge 2019 segmentation task[M]//Lecture Notes in Computer Science, vol. 11766. Berlin, Germany: Springer, 2020: 231-241.
|
| [12] |
Chen C, Liu X, Ding M, et al. 3D dilated multi-fiber network for real-time brain tumor segmentation in MRI[M]//Lecture Notes in Computer Science, vol. 11766. Berlin, Germany: Springer, 2019: 184-192.
|
| [13] |
Luo Z R, Jia Z D, Yuan Z M, et al. HDC-Net: Hierarchical decoupled convolution network for brain tumor segmentation[J]. IEEE Journal of Biomedical and Health Informatics, 2021, 25(3): 737-745. DOI:10.1109/JBHI.2020.2998146 |
| [14] |
Ma N N, Zhang X Y, Zheng H T, et al. ShuffleNet V2: Practical guidelines for efficient CNN architecture design[C]//European Conference on Computer Vision. Berlin, Germany: Springer, 2018, 11218: 122-138.
|
| [15] |
Lu S Y, Wang S H, Zhang Y D. A classification method for brain MRI via MobileNet and feedforward network with random weights[J]. Pattern Recognition Letters, 2020, 140: 252-260. |
| [16] |
Karthik R, Menaka R, Hariharan M. Learning distinctive filters for COVID-19 detection from chest X-ray using Shuffled residual CNN[J]. Applied Soft Computing, 2021. DOI:10.1016/j.asoc.2020.106744 |
| [17] |
Lu Z Y, Bai Y Z, Chen Y, et al. The classification of gliomas based on a pyramid dilated convolution resnet model[J]. Pattern Recognition Letters, 2020, 133: 173-179. |
| [18] |
Xiao Z Y, He K H, Liu J J, et al. Multi-view hierarchical split network for brain tumor segmentation[J]. Biomedical Signal Processing and Control, 2021. DOI:10.1016/j.bspc.2021.102897 |
| [19] |
Bakas S, Akbari H, Sotiras A, et al. Data descriptor: Advancing the cancer genome atlas glioma MRI collections with expert segmentation labels and radiomic features[J]. Nature Scientific Data, 2017. DOI:10.1038/sdata.2017.117 |
| [20] |
Nuechterlein N, Mehta S. 3D-ESPNet with pyramidal refinement for volumetric brain tumor image segmentation[M]//Lecture Notes in Computer Science, vol. 11384. Berlin, Germany: Springer, 2018: 245-253.
|
| [21] |
Mckinley R, Rebsamen M, Meier R, et al. Triplanar ensemble of 3D-to-2D cnns with label-uncertainty for brain tumor segmentation[M]//Lecture Notes in Computer Science, vol. 10379. Berlin, Germany: Springer, 2020: 379-387.
|
| [22] |
Milan D, Stijn B, Karel D, et al. Automated MRI based pipeline for segmentation and prediction of grade, IDH mutation and 1p19q co-deletion in glioma[J]. Computerized Medical Imaging and Graphics, 2021. DOI:10.1016/j.compmedimag.2020.101831 |



